Le modèle neutraliste de l'évolution moléculaire : La neutralité des mutations
 Jusqu’au début des années 60, le microcosme évolutionniste semblait avoir adopté, sous forme de consensus, la théorie synthétique de l’évolution qui donne une place centrale, voire unique, à la sélection naturelle, d’où son nom de « néodarwinisme ». Selon ce modèle, chaque trait d’histoire naturelle d’une espèce, anatomique, morphologique ou encore comportemental, peut être interprété en terme d’adaptation. Au plan génétique, l’hétérozygotie, c’est-à-dire l’existence de deux allèles différents d’un même gène sur les deux chromosomes homologues, était expliquée par plusieurs mécanismes. On pouvait faire intervenir l’existence, non démontrée, d’une interaction entre gènes plus ou moins voisins ayant pour effet de conférer un avantage à cette association. On parlait alors d’épistasie ou renforcement interlocus. Un autre
Jusqu’au début des années 60, le microcosme évolutionniste semblait avoir adopté, sous forme de consensus, la théorie synthétique de l’évolution qui donne une place centrale, voire unique, à la sélection naturelle, d’où son nom de « néodarwinisme ». Selon ce modèle, chaque trait d’histoire naturelle d’une espèce, anatomique, morphologique ou encore comportemental, peut être interprété en terme d’adaptation. Au plan génétique, l’hétérozygotie, c’est-à-dire l’existence de deux allèles différents d’un même gène sur les deux chromosomes homologues, était expliquée par plusieurs mécanismes. On pouvait faire intervenir l’existence, non démontrée, d’une interaction entre gènes plus ou moins voisins ayant pour effet de conférer un avantage à cette association. On parlait alors d’épistasie ou renforcement interlocus. Un autre
mécanisme invoqué était au contraire l’existence d’un renforcement intralocus qualifié d’hétérosis, de surdominance ou encore de vigueur hybride. Cette situation suppose que la valeur sélective des individus hétérozygotes soit supérieure à celle des homozygotes. Enfin, l’avantage du « rare » était envisagé comme un facteur permettant aux porteurs de caractères, et donc d’allèles, peu fréquents de tirer parti de cette rareté pour trouver des partenaires et de pouvoir ainsi transmettre les allèles en question à la génération suivante. Il faut noter que ces différentes hypothèses sont étayées par des calculs fondés sur la loi de Hardy-Weinberg et ses postulats. Le polymorphisme, c’est-à-dire l’existence de plusieurs allèles d’un même gène au sein des populations, était quant à lui assez mal documenté et ne venait pas perturber le consensus.
Dès le milieu des années 60, le développement de la technique d’électrophorèse a permis la mise en évidence d’un polymorphisme enzymatique très important au sein des populations naturelles. Deux descripteurs sont utilisés pour estimer ce polymorphisme : le taux d’hétérozygotie ou proportion moyenne de loci hétérozygotes par individus et le taux de polymorphisme ou proportion de loci polymorphes dans la population. Un certain nombre d’études proposent des valeurs pour différents groupes zoologiques : 12% d’hétérozygotie moyenne par locus et environ 30% de loci polymorphes chez Drosophila pseudoobscur a, 9% et 27% respectivement pour la souris (Britton-Davidian 1985), 7% et 28% respectivement chez l’homme (Lucotte 1983). Ces valeurs sont sous-estimées car la technique ne sépare pas deux protéines différentes de même charge et ne permet pas de distinguer deux allèles codant pour la même protéine du fait de la redondance du code génétique. Pour les autres groupes animaux et végétaux, Lucotte (1978) donne des valeurs très similaires.
Quoi qu’il en soit, le maintien d’une telle variabilité par sélection naturelle entraîne un coût reproductif ou fardeau génétique, c’est-à-dire l’élimination, à chaque génération, d’une certaine proportion des descendants, du fait de la variation de valeur sélective due aux mutations. Des calculs (pas trop simples) montrent que ce coût de la sélection naturelle est incroyablement élevé. Nous voudrions juste développer un exemple pour illustrer ce fait.
L’existence d’un fardeau de ségrégation pour des allèles sur dominants provient du fait qu’à chaque génération apparaissent des individus homozygotes dont la valeur sélective est plus faible que celle des hétérozygotes. Pour un locus biallélique donné, si on appelle 1 -si, 1, î-s2 les valeurs sélectives respectives des génotypes A1A1, A]A2 et A2A2 avec s1 > 0 et s2 > 0, le fardeau de ségrégation dû à ce seul locus polymorphe est égal à s1 s2/(‘s1 + s2) (pour le détail des calculs, voir Broussal & Viaud 1978 et Lucotte 1983). En admettant, pour simplifier, que les coefficients de sélection contre les deux génotypes homozygotes sont identiques [sx = s2 = s), on a une valeur que l’on peut écrire s/2. Si la surdominance concerne n loci, et si l’on suppose que tous les coefficients de sélection sont à peu près identiques, le fardeau de ségrégation devient alors égal 1-e~sn2 (Kimura 1990). Une petite application numérique utilisant des valeurs raisonnables pour s et n (s = 0,01 et n- 2000) nous donne un fardeau de ségrégation égal à 1-e »10 = 0,9999546. Cela signifie que chaque individu doit produire 22 000 descendants pour que la population garde un effectif constant. C’est inimaginable pour la plupart des espèces de Vertébrés. Les généticiens sélectionnistes parlent à ce propos d’un « paradoxe du fardeau de ségrégation » (Lucotte 1983) et s’en tiennent là.
Pour résoudre ce paradoxe et pour expliquer le polymorphisme observé, le généticien des populations M. Kimura, ainsi que d’autres après lui, a proposé l’hypothèse d’une dérive aléatoire de mutations neutres ou presque neutres. Cette hypothèse a été baptisée Théorie Neutraliste de l’Évolution Moléculaire (Kimura 1980, 1990). Elle part du postulat suivant : la grande majorité des mutations sont neutres du point de vue de la sélection naturelle et dérivent aléatoirement jusqu’à fixation ou élimination . Le polymorphisme observé au temps t dans une population, n’est que la vision instantanée de cette dérive touchant plusieurs loci. Ce postulat est remarquablement simple mais s’accompagne d’une mathématique relativement élaborée (pour un naturaliste, évidemment !).
La théorie neutraliste de l’évolution moléculaire n’est pas incompatible avec l’existence d’une sélection naturelle dans la mesure ou elle ne prétend pas que toutes les mutations sont neutres.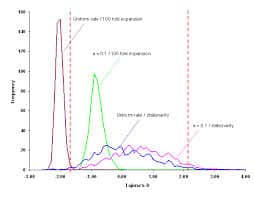 Il est dit clairement en effet que la neutralité d’une mutation dépend de la contrainte fonctionnelle de la molécule ou région de molécule qu’elle touche. Il est en effet facile de comprendre que toutes les parties d’une protéine n’ont pas la même importance pour sa fonction, le site catalytique d’une enzyme par exemple est assez localisé. La présence d’une mutation dans la région du site actif aura donc plus de conséquences qu’une mutation de même nature située dans une autre région. Cette neutralité dépend également de la localisation des nucléotides affectés (troisième lettre du codon, séquence précurseur, introns, pseudogènes…).
Il est dit clairement en effet que la neutralité d’une mutation dépend de la contrainte fonctionnelle de la molécule ou région de molécule qu’elle touche. Il est en effet facile de comprendre que toutes les parties d’une protéine n’ont pas la même importance pour sa fonction, le site catalytique d’une enzyme par exemple est assez localisé. La présence d’une mutation dans la région du site actif aura donc plus de conséquences qu’une mutation de même nature située dans une autre région. Cette neutralité dépend également de la localisation des nucléotides affectés (troisième lettre du codon, séquence précurseur, introns, pseudogènes…).